仮説生成のための発見型論文探索AIシステム
● 創薬研究者が仮説生成のために開発したAIシステム
● キーワード検索では発見できない、関連性の高い論文を抽出
● 非連続的発見のアプローチにより、セレンディピティ(予想外の発見)を体系的に実現
KIBIT Amanogawa は、非連続的発見という新しいアプローチによって、“未報告”、つまり論文に記載されていない関連性を発見する、独自の自然言語処理技術を用いた論文探索AIシステムです。検索窓に単語や文章、仮説を入力すると、AI「KIBIT」が PubMed*1に掲載されている膨大な論文情報から文脈を読み取り、未報告の関連をも予測し、関連性の高い論文を即時に抽出・解析します。KIBITによる医学・薬学研究の客観的・網羅的な解析の実現とともに、従来のキーワード検索では発見できなかった情報を見つけることが可能となり、研究者はキーワードによらない関連性の高い論文の網羅的な探索や予想外の発見、気づきから新たな着想を得ることができます。企業・機関が独自に保有するデータを読み込ませるなどのカスタマイズにも対応する、拡張性の高いシステムです。(特許登録番号:特許第6976537号)
*1 米国国立医学図書館の国立生物科学情報センターが運営する生物医学領域の論文データベース。https://pubmed.ncbi.nlm.nih.gov
「KIBIT Amanogawa」の特徴
■ アンバイアス(Unbiasedness):客観的・網羅的に解析
膨大な論文情報を、特定の研究者の知識・関心や掲載ジャーナルのインパクトファクターなどの影響を受けることなく、KIBITが客観的・網羅的に解析・抽出します。
■ セレンディピティ(Serendipity):予想外の発見を体系的に実現
非連続的発見のアプローチにより、検索ワード・文章が論文内に記載されていなくても関連性の高い論文を抽出します抽出。また、「D/G Search」では、疾患や遺伝子名を入力すると、関連性の高い疾患・遺伝子を確認でき、異なる視点から関連性のある新しい情報へのアクセスも効率的に行えます。
■ ディスカバリ(Discovery):新たな着想
KIBIT Amanogawaでの発見や気づきから新たな着想を得て、探索や研究を飛躍させることができます。
「KIBIT Amanogawa」の機能
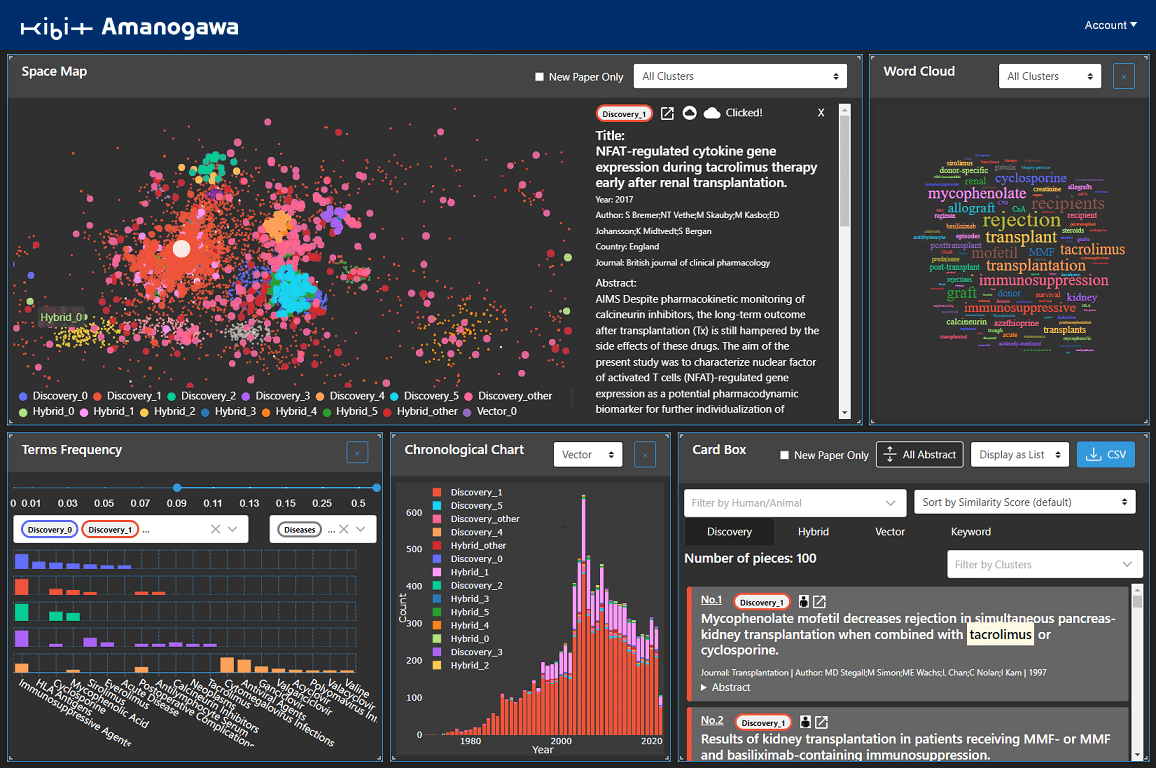
論文を独自のアルゴリズムに基づいた位置情報でプロットし、「Space Map」に表示。マップの一部を選択し、その部分のクラスタのみを対象に絞り込んだ探索や、システム画面上でのアブストラクト(要旨)の参照も可能です。データベースの論文は2週間ごとにアップデートされ、新着論文のフィルタリング機能により、新たな論文の発表状況・内容の確実なキャッチアップをサポートします。
検索結果は、さらに各クラスタがどのようなMeSH termで構成されているかを示す「Terms Frequency」、各クラスタの年次推移を示し論文内容のトレンドを一目で把握できる「Chronological Chart」などにより、様々な角度から解析可能です。「Word Cloud」は冠詞などの一般語を排除し、分子名や疾患名など、研究上の意味を持つ単語のみで構成。一般語のノイズなしに、クラスタなどの内容を把握しやすくなっています。「Card Box」では、プロットされた論文情報を検索するワードや文章との関連性の高いものから順に表示するほか、上位100報*2の論文リストをExcel形式で出力することが可能です。各パネルの表示サイズ・位置は、ユーザーが自身の好みや目的に合わせて自由にカスタマイズできます。「D/G Search」は、疾患↔遺伝子間の関連性を確認できる機能です。疾患や遺伝子名を入力すると、関連性の高い疾患・遺伝子を確認することが可能です。
なお、クラウドサービスの場合には、申し込みから最短で約1週間で利用が開始できます。
*2 Keyword検索結果、Vector検索結果、KeywordとVectorのHybrid検索結果、Vector検索結果からKeyword検索結果との重複分を除いたDiscovery検索結果に基づく各100報、計400報
ライフサイエンスAI事業本部へのお問い合わせ


